为什么要进行染色体畸变检测?
我国出生缺陷的发生率约为5.6%[1] ,其中染色体畸变约占出生缺陷遗传病因的80%以上[2],具体包括染色体数目异常、大片段缺失/重复及致病性基因组拷贝数变异(pathogenic Copy Number Variations, pCNVs)等。因此,对染色体畸变进行及时、准确的检测及评估,将有利于减少活产儿的严重出生缺陷,对于优生优育具有极为重要的临床意义。

CMA-seq:中深度全基因组测序更贴合临床所需

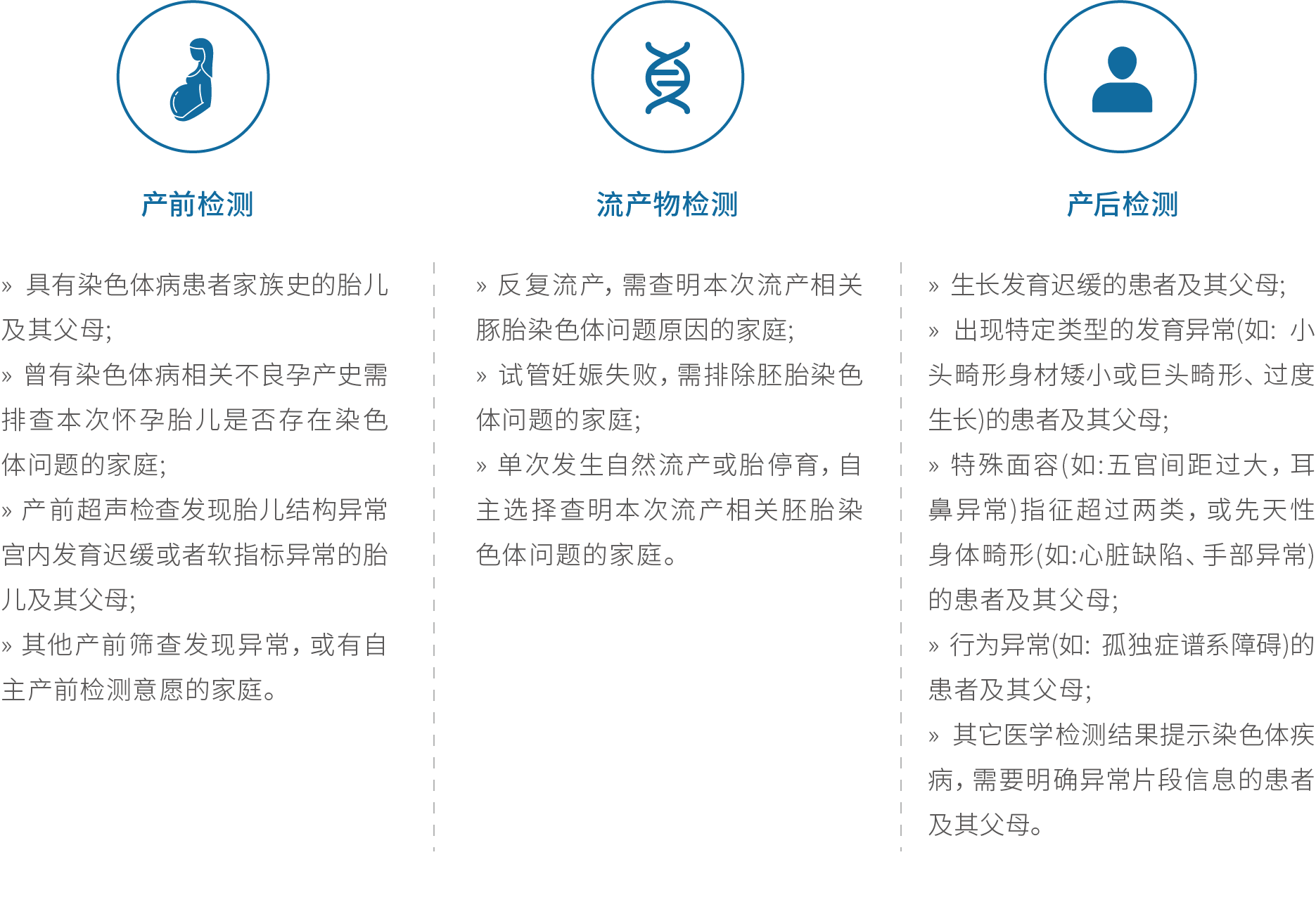
临床应用场景
常见染色体畸变评估的技术解决方案

CMA-seq技术优势与应用潜力

样本要求
血液
外周血:不低于2mL;无凝血、溶血、脂血等
①一次性无菌EDTA抗凝真空采血管(紫色帽):
2-8℃保存,一周内送达;
-20℃保存,三个月内送达(保存期间避免反复冻融);
-80℃保存,一年内送达(保存期间避免反复冻融)。
容器无破损、泄漏,采用封口膜对样本管口进行单独保护,储存容器外包装用气泡膜缓冲,防止运输碰撞破裂。
脐血:不低于1mL;无凝血、溶血、脂血等
①一次性无菌EDTA抗凝真空采血管(紫色帽):
2-8℃保存,一周内送检;
-20℃保存,三个月内送检(保存期间避免反复冻融);
-80℃保存,一年内送检(保存期间避免反复冻融)。
容器无破损、泄漏,采用封口膜对样本管口进行单独保护,储存容器外包装用气泡膜缓冲,防止运输碰撞破裂。
组织样本
羊水:5-7mL;新鲜抽取,样本清亮,无母血污染
①一次性无菌离心管:
2-8℃保存,一天内送达;
-20℃保存,一周内送达;
干冰保存,72小时内送达。
容器无破损、泄漏,采用封口膜对样本管口进行单独保护,储存容器外包装用气泡膜缓冲,防止运输碰撞破裂。
流产组织、绒毛:新鲜组织约1颗黄豆大小;绒毛2-3根(每根长度≥1.5cm)
*不接收用福尔马林、酒精、丙酮等固定液固定过的样品。组织表面不能残留血污或腐败物,请使用生理盐水充分清洗
①使用专门的组织保存管(无菌):
干冰保存,72h内送达;
2-8℃保存,2h内送达。
②使用本公司提供的组织保存液:
2-8℃保存,一个月内送达;
-20℃保存,三个月内送达(期间避免反复冻融)。
容器无破损、泄漏,采用封口膜对样本管口进行单独保护,储存容器外包装用气泡膜缓冲,防止运输碰撞破裂。
DNA
总质量:产前不低于200ng,产后不低于2μg
*浓度:在Qubit检测下浓度大于50ng/μL,电泳质检未见DNA降解
①一次性无菌、无DNA酶、无RNA酶的1.5mL离心管:
2-8℃保存,一周内送达;
-20℃保存,三个月内送达(保存期间避免反复冻融);
-80℃保存,一年内送达(保存期间避免反复冻融)。
容器无破损、泄漏,采用封口膜对样本管口进行单独保护,储存容器外包装用气泡膜缓冲,防止运输碰撞破裂。
如有疑问请致电 400-179-2998
技术·精心、医院·放心、医生·舒心、患者·安心
技术·精心
•变异类型覆盖全面
•片段拷贝分辨细致
•断点坐标定位细致
•嵌合比例估算优化
医院·放心
•国产医疗器械三类注册
•高拓展性技术平台配套
•合规化本地化配套院内开展
医生·舒心
•国产自主开发分析软件和报告系统
•编码区和非编码区的最新解读标准
患者·安心
•高性价比检测方案
参考资料
